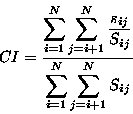


Next: 11. Théorie et pratique
Up: No Title
Previous: 9. Histoire évolutive des
Sous-sections
10. Structure secondaire de la sous-unité du
nAChR
[LE NOVÈRE N, CORRINGER PJ,
CHANGEUX JP (1998). Improved secondary structure predictions for
a nicotinic receptor subunit. Incorporation of solvent accessibility and
experimental data into a 2D representation . Biophysical Journal, 76 : 2329-2345.
]
En dépit de leurs propriétés pharmacologiques différentes,
les LGIC possèdent une structure quaternaire similaire
[182,95,230,30]. Le site composite de
liaison des ligands apparaît conservé à travers toute la superfamille. En
effet, il a été montré que le site de liaison de l'ACh, du GABA,
des benzodiazépines et de la glycine sont homologues
[294,328], revue dans
[122]10.1.
La cryo-microscopie électronique des récepteurs de l'organe électrique de
torpille a fourni des images 3D du nAChR avec une résolution de 9 Å
[325]. Une telle résolution est trop faible pour résoudre la
position spatiale et l'assignation structurale d'aucun acide aminé
particulier. Bien que le domaine extracellulaire ai été produit sous une
forme soluble [345], les quantités obtenues sont insuffisantes
pour permettre la production de cristaux utilisables pour la diffraction
des rayons X. L'approche par RMN s'est limitée à de petits
fragments [12]. Quelques tentatives ont été faites avec
d'autres méthodes comme la microscopie à force atomique [179],
mais avec une résolution inférieure à celle de la microscopie électronique
sur cristaux 2D.
Il est donc intéressant d'obtenir des informations sur l'organisation du
récepteur à partir des données actuellement disponibles, à savoir les
séquences des sous-unités. En conséquence, en parallèle aux approches
«expérimentales», des efforts ont été faits pour prédire la structure
d'une sous-unité par des techniques informatiques. Deux approches ont été
utilisées.
Les techniques de modelage comparatif ambitionnent de fournir la
description 3D d'une protéine de structure inconnue, sous réserve qu'un
modèle plausible puisse être identifié dont on connaisse la structure. Le
problème réside dans l'identification, à partir de la séquence seule, d'un
gabarit utilisable. Cependant le manque de similarité de séquence entre
les sous-unités de nAChR et n'importe quelle protéine de structure
connue oblige à utiliser les méthodes de fold recognition. Les
tests ont montré que ces méthodes sont faiblement efficaces en l'absence
totale de similitude de séquences [277]. Ces approches
souffrent également du fait qu'un gabarit 3D plausible pourrait ne pas
exister dans les banques de structures actuelles [208]. En
fait, les deux modèles proposés jusqu'à maintenant sont totalement
différents [140,322].
En parallèle, des prédictions ab initio de structure secondaire ont
été réalisées à l'aide d'algorithmes de 1ère
génération, c.-à-d.
basés sur l'analyse des acides aminés pris isolément, et ayant une
exactitude de 50-60 %, [105,243]. Afin de prendre en
compte la richesse d'information dérivée du clonage des sous-unités
homologues, j'ai réalisé une prédiction de la structure secondaire de la
sous-unité de nAChR à l'aide d'algorithmes de 3ème
génération,
c.-à-d. basés sur des alignements multiples, analysant les interactions
locales entre acides aminés, et atteignant une exactitude supérieure à
70 %. Il a été montré, pour les algorithmes de 1ère
et 2nde
génération, que la combinaison de plusieurs algorithmes indépendants
accroissait la justesse des prédictions
[235,27,368].
Je décris ici un programme qui intègre les résultats de plusieurs
algorithmes de prédiction appliqués à de multiples protéines homologues.
J'ai appliqué ce programme aux différents membres de la famille du nAChR
et de la superfamille des LGIC, afin d'accroître le rapport
signal/bruit. En sus le programme fournit les consensus de la prédiction
de l'accessibilité au solvant et de la topologie. Utilisant ces données en
combinaison avec les informations en provenance de source expérimentales,
je propose une représentation 2D d'une sous-unité typique de nAChR.
Des travaux antérieurs ont montré que l'exactitude des prédictions de
structure secondaire augmente quand on combine plusieurs algorithmes
indépendants [235,27,368]. Ici j'ai combiné les
prédictions fournies par plusieurs algorithmes de 3ème
génération,
utilisant l'information fournie par un ensemble de séquences homologues
alignées afin de prédire la structure secondaire des sous-unités du
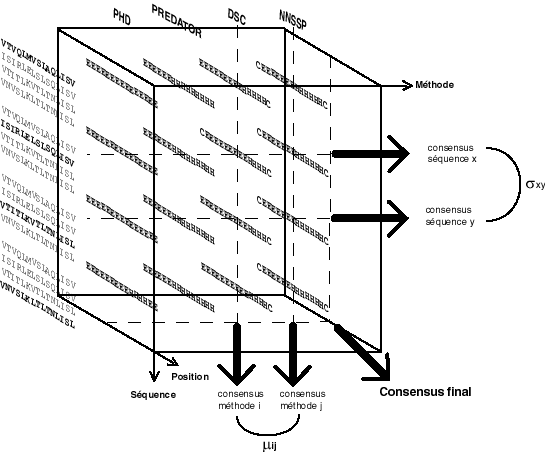
nAChR. Les programmes ont été choisis selon trois critères:
- 1.
- Ils analysent des alignements multiples au lieu de séquences uniques,
- 2.
- Ils possèdent une exactitude supérieure à 70 % pour
des prédictions à trois états (H, E, C) quand ils sont testés sur des
ensembles de protéines de structure connue, ayant des identités de
séquences inférieures à 25 % [275] ou durant des tests «en
aveugle» [270,171]
- 3.
- Chacun de ces algorithmes est fondé sur une approche prédictive
différente des autres.
Chaque programme a été appliqué successivement à chaque séquence
des alignements afin d'augmenter le rapport signal sur bruit.
10.2 Matériel et méthodes
Toutes les séquences utilisées dans cette étude peuvent être trouvées dans
la Ligand Gated Ion Channel Database à l'URL:
https://lenoverelab.org/LGICdb/LGICdb.php, voir le chapitre
2. Pour les prédictions de structures secondaires, j'ai
réalisé deux alignements multiples avec le programme
CLUSTALX [316] (disponible à l'adresse
ftp-igbmc.u-strasbg.fr) (pairwise gap opening: 10,
pairwise gap extension: 0,1, multiple gap opening: 5,
multiple gap extension: 0,05, série de matrices Blossum). Un des
alignements comporte 18 sous-unités de canaux cationiques (AL1). AL1
contient 5-HT3 de souris, les sous-unitées du nAChR  1
de torpille,
1
de torpille,  2-6,
2-6,  9 et
9 et  2-4 de rat,
2-4 de rat,  7-8
de poulet,
7-8
de poulet,
 1,
1, ,
,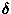 ,
,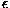 ,
de souris (un exemplaire
de chaque paralogue) et DEG3 de Cænorhabditis (Qui n'a
toujours pas d'orthologue identifié chez les vertébrés). Un autre
alignement a été construit avec 38 séquences de LGIC (cationique
et anionique) (AL2). AL2 contient les séquences présentes dans AL1 plus
GABA
,
de souris (un exemplaire
de chaque paralogue) et DEG3 de Cænorhabditis (Qui n'a
toujours pas d'orthologue identifié chez les vertébrés). Un autre
alignement a été construit avec 38 séquences de LGIC (cationique
et anionique) (AL2). AL2 contient les séquences présentes dans AL1 plus
GABA  1-6,
1-6,  1-3,
1-3, 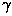 1-3,
1-3, 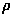 1-3,
1-3,
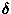 ,
glycine
,
glycine  1-3,
1-3,  de rat. Le but était de déterminer
si l'incorporation d'information venant de séquences plus distantes
améliorerait la prédiction. Une seule séquence par groupe d'orthologie a
été utilisée à cause de la forte similarité entre orthologues (et donc de
l'absence d'information additionnelle résultant de l'utilisation de
plusieurs orthologues). Le programme ASSP [282]
(disponible à l'adresse ftp://geoff.biop.ox.ac.uk/programs/assp/)
nous permet d'attendre dans le cas d'une prédiction parfaite un Q3
(c.-à-d. un pourcentage d'identité à trois états entre la prédiction et la
réalité) compris dans l'intervalle [83,45 %-100 %] pour AL1 et
[82,74 %-100 %] pour AL2. Afin d'étudier la conservation de séquence à
chaque position le long de la séquence, j'ai construit un troisième
alignement multiple à partir des séquences de 152 sous-unités. Toutes ces
séquences correspondent à des sous-unités dont on a montré qu'elles
étaient présentes dans des récepteurs (éliminant par là les membres
putatifs de la superfamille venant des projets génomes à grande échelle).
de rat. Le but était de déterminer
si l'incorporation d'information venant de séquences plus distantes
améliorerait la prédiction. Une seule séquence par groupe d'orthologie a
été utilisée à cause de la forte similarité entre orthologues (et donc de
l'absence d'information additionnelle résultant de l'utilisation de
plusieurs orthologues). Le programme ASSP [282]
(disponible à l'adresse ftp://geoff.biop.ox.ac.uk/programs/assp/)
nous permet d'attendre dans le cas d'une prédiction parfaite un Q3
(c.-à-d. un pourcentage d'identité à trois états entre la prédiction et la
réalité) compris dans l'intervalle [83,45 %-100 %] pour AL1 et
[82,74 %-100 %] pour AL2. Afin d'étudier la conservation de séquence à
chaque position le long de la séquence, j'ai construit un troisième
alignement multiple à partir des séquences de 152 sous-unités. Toutes ces
séquences correspondent à des sous-unités dont on a montré qu'elles
étaient présentes dans des récepteurs (éliminant par là les membres
putatifs de la superfamille venant des projets génomes à grande échelle).
J'ai écrit un programme en C afin d'intégrer les prédictions de
structure secondaire basées sur différents algorithmes.
SSPCA (pour Secondary Structure Prediction by
Consensus Average) a été conçu pour combiner les prédictions à
trois états et leurs probabilités venant de plusieurs programmes
appliqués à plusieurs séquences [Figure 10.1].
Figure 10.1 :
Schéma décrivant le mode de fonctionnement de
SSPCA. Seule quatre séquences sont figurées (la portion
de séquence correspond au brin- E1.
E1.
 |
Le programme SSPCA est également
capable de traiter d'autres types de prédictions comme
l'accessibilité au solvant et la topologie des protéines
membranaires. Les prédictions individuelles ne sont pas pondérées
par les similitudes de séquence. En entrée, SSPCA prend
un alignement de séquences protéiques (dans un format
CLUSTAL) et un fichier contenant les prédictions. Le
fichier de prédiction contient pour chaque séquence et pour chaque
méthode (si c'est disponible) la probabilité pour
l'hélice- ,
le brin-
,
le brin- et la boucle [0-9], la
prédiction résultante (H(elix) or E(xtended) or
C(oil)), la probabilité d'accessibilité au solvant [0-9],
l'accessibilité au solvant résultante (e(xposed) or
b(uried)), et la topologie par rapport à la membrane
(o(utside), i(nside), T(ransmembrane)).
La sortie de SSPCA est composée de (les points 1-5
concernent seulement la prédiction de structure secondaire):
et la boucle [0-9], la
prédiction résultante (H(elix) or E(xtended) or
C(oil)), la probabilité d'accessibilité au solvant [0-9],
l'accessibilité au solvant résultante (e(xposed) or
b(uried)), et la topologie par rapport à la membrane
(o(utside), i(nside), T(ransmembrane)).
La sortie de SSPCA est composée de (les points 1-5
concernent seulement la prédiction de structure secondaire):
- 1.
- les
M x S
prédictions
P(mi,sx) - où M est le nombre de méthodes,
S le nombre de séquences, mi est la ième
méthode et sx la xème
séquence - projetées sur l'alignement (insertion d'une délétion
dans les prédictions quand il y en a une dans l'alignement). Chaque
P(mi,sx) est donc une chaîne de caractères de longueur
identique à l'alignement, chaque caractère
 .
.
- 2.
- Les
M x (M x S - 1)/2�
comparaisons par paire
C[P(mi,sx),P(mj,sy)] des
prédictions
P(mi,sx) et
P(mj,sy). Si
 est
l'ensemble des positions de l'alignement où ni
P(mi,sx) ni
P(mj,sy) ne contiennent de délétion (Gap), c.-à-d., où les deux prédictions
sont définies,
est
l'ensemble des positions de l'alignement où ni
P(mi,sx) ni
P(mj,sy) ne contiennent de délétion (Gap), c.-à-d., où les deux prédictions
sont définies,
où
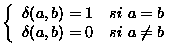 (
(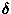 de Kronecker)
de Kronecker)
et card(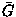 )
est le cardinal (le nombre d'éléments) de
)
est le cardinal (le nombre d'éléments) de 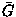 .
.
- 3.
- La congruence entre méthodes µi,j :
c'est le pourcentage
d'identité entre les consensus, toutes séquences
confondues, de deux méthodes.
pour chaque paire (i,j), i 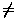 j. Ce paramètre
permet d'évaluer la variabilité entre méthodes et donc la
fiabilité des prédictions.
j. Ce paramètre
permet d'évaluer la variabilité entre méthodes et donc la
fiabilité des prédictions.
- 4.
- La congruence entre séquences
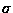 x,y :
c'est le pourcentage d'identité entre les consensus, toutes
méthodes confondues, de deux séquences.
x,y :
c'est le pourcentage d'identité entre les consensus, toutes
méthodes confondues, de deux séquences.
pour chaque paire (x,y), x 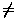 y. Ce
paramètre permet d'évaluer l'impact de la variation de séquence
au sein du groupe sur la prédiction.
y. Ce
paramètre permet d'évaluer l'impact de la variation de séquence
au sein du groupe sur la prédiction.
- 5.
- Les prédictions consensus et la somme des
probabilités : par séquences, par méthodes, et in toto
(ainsi que le pourcentage d'hélice-
 et de feuillet-
et de feuillet- pour
chaque consensus de prédiction). Pour chaque position, le consensus
est calculé comme l'état majoritaire. Dans le cas de cardinaux
identiques, la priorité arbitraire est E>H>C>`-'. Le pourcentage
d'hélice-
pour
chaque consensus de prédiction). Pour chaque position, le consensus
est calculé comme l'état majoritaire. Dans le cas de cardinaux
identiques, la priorité arbitraire est E>H>C>`-'. Le pourcentage
d'hélice- et de feuillet-
et de feuillet- est donné par rapport à
la longueur totale du consensus sans délétion.
est donné par rapport à
la longueur totale du consensus sans délétion.
- 6.
- Le consensus d'accessibilité au solvant.
Dans le cas de cardinaux identiques, la priorité arbitraire est
b>e>`-'
- 7.
- La topologie consensus.
Dans le cas de cardinaux identiques, la priorité arbitraire est
T>i>o>`-'
Le programme [273,274,275] est composé de
plusieurs réseaux de neurones en cascade (préalablement entraînés sur des
protéines de structure connue). Un premier réseau prend en entrée un
ensemble de vecteurs représentant la composition en acides aminés à chaque
position d'une fenêtre glissant le long d'un alignement multiple. Sa
sortie est composée d'un vecteur représentant les probabilités pour chacun
des trois états du résidu au centre de la fenêtre. Comme la structure
secondaire d'un résidu n'est pas indépendante de celle de ses voisins, un
second réseau prend en compte ces interactions locales. Il prend en entrée
les vecteurs présents dans une fenêtre glissant le long de la sortie
précédente. Sa propre sortie est un vecteur de probabilités affinées. Une
autre étape consiste à moyenner (pour chaque état) les sorties de
plusieurs réseaux entraînés indépendamment les uns des autres. Finalement
une décision winner-takes-all assigne l'état structural au résidu.
Aucune règle explicite n'est incluse dans l'algorithme. PHD peut
générer son propre alignement à partir de la séquence soumise (à l'aide du
programme MAXHOM [288]). En conséquence, pour chaque
séquence de AL1 et AL2, un alignement différent a été fabriqué à partir
des banques de données publiques et utilisé pour la prédiction.
PHD est accessible à l'URL:
http://www.embl-heidelberg.de/predictprotein/predictprotein.html.
Le programme [113,114] est basé sur le calcul des
propensions de chacune des 400 paires d'acides aminés à interagir par
l'intermédiaire d'un des types de liaison  ou bien à l'intérieur
d'une hélice-
ou bien à l'intérieur
d'une hélice- Il incorpore donc des statistiques sur les
interactions non-locales. predator utilise également des
propensions pour chacun des trois états dérivées d'une approche de
nearest-neighbor (voir plus loin). Afin d'utiliser l'information en
provenance des protéines homologues, predator s'appuie sur des
alignement locaux par paire. predator peut utiliser des
alignements Clustal comme entrée. predator est
accessible aux urls:
http://www.embl-heidelberg.de/cgi/predator_serv.pl et
http://bioweb.pasteur.fr/seqanal/interfaces/predator.html.
Le programme [171] combine plusieurs paramètres
explicites afin de produire une prédiction porteuse d'une
«signification» biologique. Il lance le programme
GORIII [131] (basé sur la théorie de l'information
appliquée aux interactions locales) sur chaque séquence pour obtenir des
potentiels moyens pour les trois états. En sus, DSC utilise la
présence d'insertions/délétions, la distance à partir de l'extrémité de la
chaîne, le moment de conservation et le moment d'hydrophobie (les derniers
paramètres calculés dans le cas d'une hélice-
Il incorpore donc des statistiques sur les
interactions non-locales. predator utilise également des
propensions pour chacun des trois états dérivées d'une approche de
nearest-neighbor (voir plus loin). Afin d'utiliser l'information en
provenance des protéines homologues, predator s'appuie sur des
alignement locaux par paire. predator peut utiliser des
alignements Clustal comme entrée. predator est
accessible aux urls:
http://www.embl-heidelberg.de/cgi/predator_serv.pl et
http://bioweb.pasteur.fr/seqanal/interfaces/predator.html.
Le programme [171] combine plusieurs paramètres
explicites afin de produire une prédiction porteuse d'une
«signification» biologique. Il lance le programme
GORIII [131] (basé sur la théorie de l'information
appliquée aux interactions locales) sur chaque séquence pour obtenir des
potentiels moyens pour les trois états. En sus, DSC utilise la
présence d'insertions/délétions, la distance à partir de l'extrémité de la
chaîne, le moment de conservation et le moment d'hydrophobie (les derniers
paramètres calculés dans le cas d'une hélice- et d'un
brin-
et d'un
brin- ). Une combinaison linéaire de ces différents attributs
donne en sortie une prédiction qui est subséquemment filtrée à l'aide de
règles (arbitrairement ?) définies par l'auteur. Les options suivantes ontr
été utilisées: `-a' (pour stopper la suppression des parties mal alignées)
`-i' (pour stopper la suppression des singletons) '-f1' (pour appliquer
les règles de filtrage une seule fois) '-w' (alignement
Clustal(W) en entrée). dsc est accessible aux
\textsc{url}s:
http://bonsai.lif.icnet.uk/bmm/dsc/dsc_read_align.html et
http://bioweb.pasteur.fr/seqanal/interfaces/dsc.html.
Le programme [287] est basé sur un algorithme de
nearest-neighbor (parfois improprement appelée la méthode
«homologue») [194,235]. L'idée de base de
l'approche des nearest-neighbor est la prédiction de l'état
structural du résidu central d'un segment test, fondée sur la structure
secondaire de segments similaires venant de protéines dont la structure
est connue. L'information provenant de segments différents est pondérée
selon leur similitude avec le segment à prédire (définie par leur séquence
ou d'autres propriétés). NNSSP est une amélioration de
l'algorithme décrit par [362] qui sélectionne les voisins à partir
de scores d'environnement [34] et combine à l'aide d'un
perceptron (un réseau de neurones sans couche cachée) les prédictions
effectuées avec des paramètres différents (longueur des
nearest-neighbor, score d'environnement etc.). En plus de ce
dernier programme, il incorpore l'information venant d'un alignement
multiple (en moyennant les scores obtenus par chaque homologue pour
pondérer chaque voisin). NNSSP est accessible aux URLs:
http://dot.imgen.bcm.tmc.edu:9331/pssprediction/pssp.html et
http://bioweb.pasteur.fr/seqanal/interfaces/nnssp.html. J'ai écrit
un programme, clu2nnssp, pour convertir un alignement CLUSTAL en
alignement NNSSP. Il est disponible à l'URL:
http://www.pasteur.fr/units/neubiomol/softwares.html.
Le programme [276] calcule la probabilité d'accessibilité au
solvant. Comme PHDsec, ce programme est basé sur un système de réseaux de
neurones. Sa justesse de prédiction pour 3 états (buried,
intermediate, exposed) n'est que de 58 %. Mais l'accessibilité au
solvant est une caractéristique peu conservée (même en cas de similitude
de séquence significative) et ce score pourrait être proche de l'optimum
(le score obtenu par le modelage par homologie est de 70 %). La sortie
utilisée par SSPCA est une sortie à 2 états.
Le programme [272,271] prédit la position des
segments transmembranaires avec une exactitude de 95 %, et leur
topologie (in
). Une combinaison linéaire de ces différents attributs
donne en sortie une prédiction qui est subséquemment filtrée à l'aide de
règles (arbitrairement ?) définies par l'auteur. Les options suivantes ontr
été utilisées: `-a' (pour stopper la suppression des parties mal alignées)
`-i' (pour stopper la suppression des singletons) '-f1' (pour appliquer
les règles de filtrage une seule fois) '-w' (alignement
Clustal(W) en entrée). dsc est accessible aux
\textsc{url}s:
http://bonsai.lif.icnet.uk/bmm/dsc/dsc_read_align.html et
http://bioweb.pasteur.fr/seqanal/interfaces/dsc.html.
Le programme [287] est basé sur un algorithme de
nearest-neighbor (parfois improprement appelée la méthode
«homologue») [194,235]. L'idée de base de
l'approche des nearest-neighbor est la prédiction de l'état
structural du résidu central d'un segment test, fondée sur la structure
secondaire de segments similaires venant de protéines dont la structure
est connue. L'information provenant de segments différents est pondérée
selon leur similitude avec le segment à prédire (définie par leur séquence
ou d'autres propriétés). NNSSP est une amélioration de
l'algorithme décrit par [362] qui sélectionne les voisins à partir
de scores d'environnement [34] et combine à l'aide d'un
perceptron (un réseau de neurones sans couche cachée) les prédictions
effectuées avec des paramètres différents (longueur des
nearest-neighbor, score d'environnement etc.). En plus de ce
dernier programme, il incorpore l'information venant d'un alignement
multiple (en moyennant les scores obtenus par chaque homologue pour
pondérer chaque voisin). NNSSP est accessible aux URLs:
http://dot.imgen.bcm.tmc.edu:9331/pssprediction/pssp.html et
http://bioweb.pasteur.fr/seqanal/interfaces/nnssp.html. J'ai écrit
un programme, clu2nnssp, pour convertir un alignement CLUSTAL en
alignement NNSSP. Il est disponible à l'URL:
http://www.pasteur.fr/units/neubiomol/softwares.html.
Le programme [276] calcule la probabilité d'accessibilité au
solvant. Comme PHDsec, ce programme est basé sur un système de réseaux de
neurones. Sa justesse de prédiction pour 3 états (buried,
intermediate, exposed) n'est que de 58 %. Mais l'accessibilité au
solvant est une caractéristique peu conservée (même en cas de similitude
de séquence significative) et ce score pourrait être proche de l'optimum
(le score obtenu par le modelage par homologie est de 70 %). La sortie
utilisée par SSPCA est une sortie à 2 états.
Le programme [272,271] prédit la position des
segments transmembranaires avec une exactitude de 95 %, et leur
topologie (in
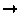 out ou le contraire) avec une justesse
de 86 %.
J'ai écrit le programme CONSINDEX pour calculer la conservation
de séquence entre séquences homologues à chaque position d'un alignement
multiple. Le programme prend en entrée un alignement au format
CLUSTAL et une matrice de similarité. Il calcule d'abord les
N(N-1)/2 similarités globales Sij (identités si la matrice identité
est entrée) des N séquences. Puis pour chaque position de l'alignement,
un index de conservation est calculé comme suit:
out ou le contraire) avec une justesse
de 86 %.
J'ai écrit le programme CONSINDEX pour calculer la conservation
de séquence entre séquences homologues à chaque position d'un alignement
multiple. Le programme prend en entrée un alignement au format
CLUSTAL et une matrice de similarité. Il calcule d'abord les
N(N-1)/2 similarités globales Sij (identités si la matrice identité
est entrée) des N séquences. Puis pour chaque position de l'alignement,
un index de conservation est calculé comme suit:
où sij est l'élément pertinent de la matrice de similarité pour les
séquences i et j à la position considérée. Dans le travail présenté
par la suite, la matrice utilisée est la matrice par défaut du programme
GAP (suite WISCONSIN du GCG
[85]). Pour ce faire elle a été ré-échelonnée de
[-1,2···1,5] à [0···100]. Le gap a été ajouté comme acide
aminé indépendant, tout élément de matrice le concernant étant nul.
CONSINDEX est disponible à l'URL:
http://www.pasteur.fr/units/neubiomol/softwares.html.
Les congruences entre méthodes µi,j
pour chaque paire de
méthodes sont listées dans le tableau 10.1.
Tableau 10.1 :
Congruence des prédictions
fournies par les différentes méthodes, µi,j�
exprimées comme
moyenne (±SD). Les valeurs hautes viennent de l'analyse d'AL1,
les valeurs basses viennent de l'analyse d'AL2.
| |
PHD |
PREDATOR |
DSC |
| PREDATOR |
73,58 (±5.85) |
|
|
| 69,32 (±6.35) |
|
|
| DSC |
73,20 (±3.69) |
67,14 (±2,39) |
|
| 66,67 (±4.7) |
57,12 (±2,98) |
|
| NNSSP |
76,64 (±4.84) |
76,44 (±3,83) |
78,07 (±2,11) |
| 71,70 (±4.42) |
60,53 (±5,45) |
69,13 (±5,05) |
Les quatre méthodes donnent des valeurs de µi,j
supérieures à
67 %. L'utilisation d'un ensemble de séquences plus important entraîne
une diminution de µ,
qui reste néanmoins au-dessus de 57 %. La
congruence entre les prédictions consensus par séquence
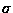 x,y
a
aussi été examinée pour chaque paire de séquences. Les prédictions pour
les sous-unités des LGIC cationiques sont cohérentes, les valeurs
de congruence variant de
x,y
a
aussi été examinée pour chaque paire de séquences. Les prédictions pour
les sous-unités des LGIC cationiques sont cohérentes, les valeurs
de congruence variant de
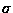 deg3,a1 = 80.8 % à
deg3,a1 = 80.8 % à
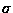 a3,a6 = 95.3 %.
Avec AL2, le plus petit
a3,a6 = 95.3 %.
Avec AL2, le plus petit 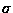 est
juste au-dessus de 64 %, une valeur largement supérieure à celle attendue
pour une prédiction aléatoire (qui est 33 % pour une comparaison à trois
états non biaisée, et 38 % si l'on prend en compte les biais existant
dans la PDB actuelle). La bonne congruence entre les différentes
prédictions pour les membres de la famille du nAChR est illustrée sur la
figure 10.2, où les pics de prédictions sont nets et où 17 des 25
éléments structuraux finals sont prédits dans plus de 90 % des cas.
est
juste au-dessus de 64 %, une valeur largement supérieure à celle attendue
pour une prédiction aléatoire (qui est 33 % pour une comparaison à trois
états non biaisée, et 38 % si l'on prend en compte les biais existant
dans la PDB actuelle). La bonne congruence entre les différentes
prédictions pour les membres de la famille du nAChR est illustrée sur la
figure 10.2, où les pics de prédictions sont nets et où 17 des 25
éléments structuraux finals sont prédits dans plus de 90 % des cas.
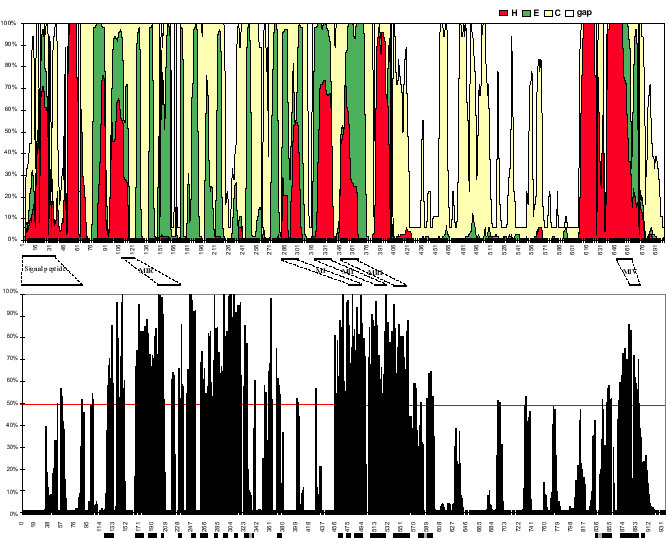
Figure 10.2 :
Graphe supérieur, incidence relative par position des trois
états dans les prédictions. Pour chaque résidu de AL1, est représenté
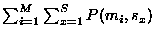 ,
la somme de toutes prédictions par toutes les méthodes
et pour toutes les séquences. Le diagramme est cumulatif, c.-à-d.
que la différence entre les hauteurs des courbes à une position
donnée sont informatives, pas les hauteurs des courbes
elles-mêmes. Graphe inférieur, index de conservation par position.
Pour chaque position d'un alignement de 152 sous-unités de
LGIC, un index de conservation a été calculé par
CONSINDEX. Les rectangles noirs au-dessous du graphe
représentent les structures finalement prédites (hélice-
,
la somme de toutes prédictions par toutes les méthodes
et pour toutes les séquences. Le diagramme est cumulatif, c.-à-d.
que la différence entre les hauteurs des courbes à une position
donnée sont informatives, pas les hauteurs des courbes
elles-mêmes. Graphe inférieur, index de conservation par position.
Pour chaque position d'un alignement de 152 sous-unités de
LGIC, un index de conservation a été calculé par
CONSINDEX. Les rectangles noirs au-dessous du graphe
représentent les structures finalement prédites (hélice- ou brin-
ou brin- )
pour la sous-unité mature. Si une hélice-
)
pour la sous-unité mature. Si une hélice- est adjacente à un brin-
est adjacente à un brin- ,
il n'y a qu'un
rectangle.
,
il n'y a qu'un
rectangle.
 |
Les prédictions consensus par séquence sont très
similaires. Les positions des structures secondaires sont à peu près
identiques, les assignations montrant peu de variation. Les prédictions
consensus par méthodes sont plus variables, restant toutefois similaires.
L'assignation des structures varie parfois, ainsi que (mais très rarement)
leur existence. La ressemblance des structures 3D est proportionnelle à
l'identité des séquences [50,109]. L'incorporation
d'information venant de séquences distantes (donc ayant une structure
légèrement différente) est supposée accroître la fiabilité des structures
prédites (si elles sont prédites, c'est qu'elles sont conservées), bien
que diminuant l'exactitude de la prédiction globale
[282,278,310]. Les valeurs de 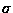 obtenues avec AL2 ont été tracées en fonction des similarités de séquences
globales déterminées par CONSINDEX [figure 10.3].
obtenues avec AL2 ont été tracées en fonction des similarités de séquences
globales déterminées par CONSINDEX [figure 10.3].
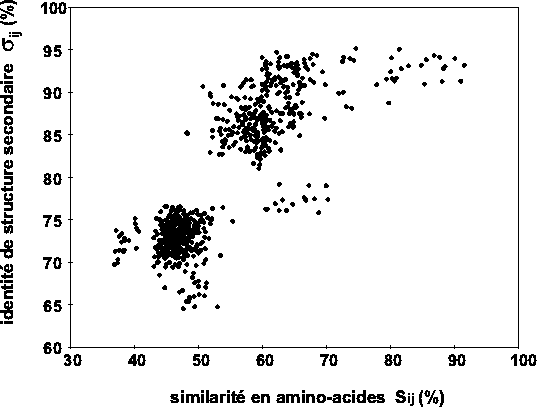
Figure 10.3 :
Corrélation entre la similarité de séquence et l'identité de
prédiction de structure secondaire. Pour N séquences, il y a
(N-1)(N-2)/2 similarités de séquences. Ici sont représentés 703 points
correspondant aux 38 séquences de AL2. Les similarités en acide-aminé
Sij ont été calculées par le programme CONSINDEX. Elles sont
reportées point par point par rapport aux identités de prédictions de
structure secondaire,
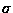 i,j�
définies au paragraphe
10.2. Chaque point est donc la comparaison d'une similarité de
séquence versus 16 similarités de prédiction (quatre méthodes de
prédiction pour chaque séquence). Une analyse de régression bivariée
montre que la corrélation est significative (n=703, r=0.882, p<0.001).
Notez les deux composantes: la concentration en bas et à gauche représente
les comparaisons anionique/cationique (p. ex. GABAA
vs. nAChR) alors que la concentration un peu moins dense en haut
et à droite représente les comparaisons anionique/anionique ou
cationique/cationique.
i,j�
définies au paragraphe
10.2. Chaque point est donc la comparaison d'une similarité de
séquence versus 16 similarités de prédiction (quatre méthodes de
prédiction pour chaque séquence). Une analyse de régression bivariée
montre que la corrélation est significative (n=703, r=0.882, p<0.001).
Notez les deux composantes: la concentration en bas et à gauche représente
les comparaisons anionique/cationique (p. ex. GABAA
vs. nAChR) alors que la concentration un peu moins dense en haut
et à droite représente les comparaisons anionique/anionique ou
cationique/cationique.
|
|
Une corrélation non-ambiguë est mise en évidence (N=703, R=0.882,
p<0.001). Deux composantes principales émergent à partir des
comparaisons: une population à faible similarité représente les
comparaisons anionique/cationique (p. ex. GABAA
vs. nAChR), et une à plus forte similarité représente les
comparaisons anionique/anionique ou cationique/cationique. Ces
données montrent que les variations entre les prédictions de
structure secondaire ne sont pas aléatoires, comme attendu en cas
d'imperfection des algorithmes de prédiction. Au contraire, elles
sont liées à la variation de séquence. Cela reflète le fait que,
si les structures formant l'échafaudage des sous-unités sont
conservées entre différents membres de la superfamille -- ce qui
est soutenu par un corpus de données important [122]
--, l'assignation structurale au niveau du résidu individuel
peut varier (p. ex. aux extrémités des éléments structuraux). Une autre
conclusion peut être dérivée de la figure 10.3: plus les
homologues utilisés seront éloignés de la séquence à prédire, moins
l'information obtenue sera fiable. Un trade-off est atteint entre
l'information gagnée dans l'utilisation d'alignements multiples (fiabilité
des positions et des assignations des éléments structuraux) et les
mauvaises prédictions au niveau des résidus individuels dues à la
divergence de séquence [282,278]. Il n'y pas de méthode
connue pour établir le meilleur compromis.
Les résultats finals obtenus avec les deux alignements AL1 et AL2 sont
très semblables, avec quelques résidus seulement prédits dans un état
différent. Toutes les structures sauf une sont prédites également avec les
deux ensembles de séquences, et dans tous ces cas, l'assignation est la
même. En conséquence, excepté quand c'est explicitement dit, les résultats
présentés plus bas sont ceux obtenus avec AL1 (voir figure
10.4).
Figure 10.4 :
Alignement des sous-unités de canaux cationiques (AL1) et prédictions
de structure 1D. La première ligne donne les limites des structures
secondaires affinées et leurs noms respectifs (lettres pour les
hélices- et chiffres pour les brins-
et chiffres pour les brins- ). Suit l'alignement,
créé par CLUSTALX. Les couleurs sont réglées selon les acides
aminés ou bien le consensus à chaque position. Par exemple, une proline
est toujours jaune alors qu'une cystéine est pourpre si c'est le résidu
consensus mais peut être bleu si le consensus est un résidu hydrophobe.
Sous l'alignement et la réglette, la prédiction consensus brute de
structure secondaire est présentée. Les boites magenta et vert
représentent les prédictions affinées. Sous la structure secondaire est
présentée la prédiction d'accessibilité au solvant. Sur la dernière ligne
est reportée la topologie consensus. Sur cette même ligne sont reportés
les résultats du marquage par affinité du site de liaison (A, B, C, D, E, F) du canal (l), la mutagenèse dans le canal (m) et la glycosylation
(N) naturelle ou induite.
). Suit l'alignement,
créé par CLUSTALX. Les couleurs sont réglées selon les acides
aminés ou bien le consensus à chaque position. Par exemple, une proline
est toujours jaune alors qu'une cystéine est pourpre si c'est le résidu
consensus mais peut être bleu si le consensus est un résidu hydrophobe.
Sous l'alignement et la réglette, la prédiction consensus brute de
structure secondaire est présentée. Les boites magenta et vert
représentent les prédictions affinées. Sous la structure secondaire est
présentée la prédiction d'accessibilité au solvant. Sur la dernière ligne
est reportée la topologie consensus. Sur cette même ligne sont reportés
les résultats du marquage par affinité du site de liaison (A, B, C, D, E, F) du canal (l), la mutagenèse dans le canal (m) et la glycosylation
(N) naturelle ou induite.
 |
Les proportions de chacun des trois états dans l'ensemble des
prédictions sont présentées pour chaque position sur le graphe supérieur de la figure
10.2. La figure 10.4 présente
la prédiction consensus brute, en texte, juste dessous l'alignement. L'indice de conservation déterminé sur l'ensemble de
la superfamille des LGIC (152 sous-unités) est tracé sur le graphe inférieur
de la figure 10.2. Dessous sont tracés les
emplacements des structures secondaires (rectangles noirs sous le
graphe). Dans tous les cas sauf trois (E9, HF
et HG)
les
structures prédites sont localisées dans des régions de haute
conservation (supérieure à 50 %). La région de E9 est en fait
hautement conservée excepté pour la sous-unité unc38 du nAChR de
nématode. Les régions de HF
et HG
sont hautement conservées
dans les sous-unités de canaux cationiques. En résumé, au sein de
la famille des canaux cationiques de vertébrés, toutes les
structures prédites sont localisées dans des régions de haute
conservation. Ce fait est important car les variations entre les
membres de la superfamille sont probablement localisées dans les
régions de séquence variable. Un élément structural prédit dans
une région conservée est donc plus fiable.
PHD, DSC, et NNSSP fournissent des probabilités
pour les trois états en sus des états finalement prédis. La combinaison de
ces probabilités permet la correction des décisions seuillées au niveau de
chaque séquence, qui peut entraîner des fausses assignations. Elle offre
la possibilité de résoudre quelques problèmes comme les singletons (résidu
structuré isolé) ou bien les acides aminés situés aux limites des motifs
de structure secondaire. Les changements effectués par cette étape de
filtrage n'affectent que 29 positions (sur 489). La prédiction affinée
résultante contient (sans compter le peptide signal) 9 hélices- (longueur moyenne 13,9 acides-aminés) désignés de HA
à HH,
et 17 brins-
(longueur moyenne 13,9 acides-aminés) désignés de HA
à HH,
et 17 brins- (longueur moyenne 6,6 acides-aminés) désignés de E1
à E17. Leurs positions et leurs longueurs sont résumés dans le
tableau 10.2.
(longueur moyenne 6,6 acides-aminés) désignés de E1
à E17. Leurs positions et leurs longueurs sont résumés dans le
tableau 10.2.
Tableau 10.2 :
Récapitulation
de la position des éléments structuraux et de leur longueur.
La position dans la séquence mature de  7 de poulet est
donnée pour des comparaisons avec d'autres travaux. Les limites
sont incluses (c.-à-d. que 113-115 signifie 113-114-115)
7 de poulet est
donnée pour des comparaisons avec d'autres travaux. Les limites
sont incluses (c.-à-d. que 113-115 signifie 113-114-115)
| hélices |
position |
brins |
position |
| A |
50-61 (Phe3-Asn14) |
1 |
78-90 (Leu28-Met40) |
| B |
97-110 (Gln47-Thr60) |
2 |
113-115 (Tyr63-Gln65) |
| C |
295-300 (Leu220-Ala225) |
3 |
125-128 (Lys75-Arg78) |
| D |
324-337 (Val245-Glu258) |
4 |
139-142 (Ile89-Tyr92) |
| E |
348-355 (Leu269-Ser276) |
5 |
151-161 (Asp100-Asn110) |
| F |
385-400 (Pro305-Leu330) |
6 |
173-175 (Cys115-Tyr117) |
| G |
609-627 (Pro408-Arg425) |
7 |
186-191 (Tyr128-Trp133) |
| H |
637-656 (Ala432-Val451) |
8 |
200-203 (Asn142-Phe145) |
| |
|
9 |
213-218 (Ser154-Met159) |
| |
|
10 |
245-249 (Trp173-Gly177) |
| |
|
11 |
272-278 (Ile197-Met203) |
| |
|
12 |
284-290 (Tyr209-Leu215) |
| |
|
13 |
301-305 (Leu226-Leu230) |
| |
|
14 |
318-323 (Thr244-Ile243) |
| |
|
15 |
356-373 (Thr277-Tyr294) |
| |
|
16 |
657-662 (Phe452-Ile457) |
| |
|
17 |
677-670 (Gly462-Met465) |
Exceptées deux larges hélices encadrant un grand brin- à
l'extrémité amino-terminale, la partie extracellulaire des sous-unités est
prédite entièrement en feuillet-
à
l'extrémité amino-terminale, la partie extracellulaire des sous-unités est
prédite entièrement en feuillet- ,
formée d'une succession de petit
brins.
,
formée d'une succession de petit
brins.
La structure de la portion carboxy-terminale de HA�
est
cohérente avec le motif d'accessibilité au solvant (décrit par la
suite comme une chaîne de `e' pour exposed et
`b' pour buried) à savoir ``bbeebbee'',
sa portion amino-terminale étant complètement exposée.
La structure au centre de E1 est également en accord avec le motif
d'accessibilité au solvant ``bebebe'', ses deux extrémités étant
prédites comme complètement cachées. La prédiction de sa partie
carboxy-terminale est moins fiable, puisque chacun des consensus de
séquence et trois des consensus de méthodes de AL1 la prédisent en
hélice- (voir figure 10.2, graphe supérieur). Une structure
en hélice-
(voir figure 10.2, graphe supérieur). Une structure
en hélice- pourrait donc être envisagée pour les quatre derniers
résidus. Cependant, dans le cas d'AL2, seul le consensus de
PREDATOR, ainsi que les consensus de
pourrait donc être envisagée pour les quatre derniers
résidus. Cependant, dans le cas d'AL2, seul le consensus de
PREDATOR, ainsi que les consensus de 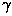 et
et  8 du
nAChR présentent quelques résidus prédits en hélice-
8 du
nAChR présentent quelques résidus prédits en hélice- .
Cet unique
pas d'hélice pourrait donc être une caractéristique spécifique des
sous-unités des familles cationiques.
.
Cet unique
pas d'hélice pourrait donc être une caractéristique spécifique des
sous-unités des familles cationiques.
La région antigénique principale (MIR pour Main
Immunogenic Region) est localisée de la fin de HB
au début de
E3 [323]. Ce segment était déjà connu pour être
exposé au solvant, puisqu'il est directement impliqué dans
plusieurs formes de la maladie auto-immune myasthenia
gravis. Effectivement, sa partie centrale est prédite totalement
accessible au solvant.
L'assignation de HB
apparaît consistante dans toutes les
prédictions exceptées celles de DSC pour AL1 et AL2 ainsi
que celle de PHD pour AL2 (cependant seuls quelques
résidus sont prédits en brin- ). Le motif d'accessibilité au
solvant est plus en accord avec un brin-
). Le motif d'accessibilité au
solvant est plus en accord avec un brin- dans la partie
carboxy-terminale.
dans la partie
carboxy-terminale.
La structure E2 (longue de 3 résidus) n'est pas prédite par
l'analyse de AL2. C'est le seul élément structural qui diffère
entre les deux analyses.
L'assignation de E3 est contredite par des expériences de
cross-linking [342] montrant que ses deux premiers
résidus exposeraient leurs chaînes latérales dans la même direction.
E4 et E7 sont prédits complètement enfouis.
E5 comme E8 sont en accord avec l'accessibilité au solvant
``ebebebeb''.
Les prédictions de E12-15 et HC-E
sont probablement
moins justes que celles des parties extra-membranaires. En effet,
les programmes de prédiction de structure secondaire n'ont pas été
écrits pour, ou testés avec, des protéines membranaires (voir le
paragraphe 10.4). La longueur des structures prédites
varie considérablement selon l'ensemble de séquences utilisé. Avec
AL2, HD
est plus court (dans MII), HE
est plus long
et E15 plus court (dans MIII).
Finalement, HF
et HG
sont pleinement compatibles avec les
prédictions d'accessibilité au solvant,
``bbebbbebbbebbbebb''
et ``eebeebbebbebbbeeb'', indiquant l'existence d'une face
exposée et d'une autre enfouie.
Les données expérimentales disponibles peuvent être ajoutées aux
prédictions 1D fournies par SSPCA. On définit ainsi une enveloppe
de contraintes structurales, qui permet de proposer un repliement 2D de la
chaîne peptidique [figure 10.5].
Figure 10.5 :
Représentation 2D d'une sous-unité typique. La
perspective est vue de l'intérieur du pore, perpendiculairement à
la membrane. Les hélices- et la longueur des brins-
et la longueur des brins- sont dessinés à l'échelle. La longueur des parties non-structurées
est approximativement à l'échelle. La double-flèche grise signifie
que les deux parties du domaine extracellulaire doivent se replier
ensembles. Les segments bleus représentent les positions des
morceaux (de longueur supérieure ou égale à 4 résidus) exposés au
solvant. Le lien jaune représente le pont di-sulfure. Les acides
aminés identifiés avec le marquage par affinité sont notés (la
numérotation est celle de
sont dessinés à l'échelle. La longueur des parties non-structurées
est approximativement à l'échelle. La double-flèche grise signifie
que les deux parties du domaine extracellulaire doivent se replier
ensembles. Les segments bleus représentent les positions des
morceaux (de longueur supérieure ou égale à 4 résidus) exposés au
solvant. Le lien jaune représente le pont di-sulfure. Les acides
aminés identifiés avec le marquage par affinité sont notés (la
numérotation est celle de  7 de poulet). Notez que ce
repliement est une représentation 2D, et n'a rien à voir avec un
modèle 3D. En effet, les brins-
7 de poulet). Notez que ce
repliement est une représentation 2D, et n'a rien à voir avec un
modèle 3D. En effet, les brins- sont placés parallèles et
dans un ordre suivant la séquence primaire uniquement pour des
raisons de facilité. La figure n'implique en aucune façon des
interactions
sont placés parallèles et
dans un ordre suivant la séquence primaire uniquement pour des
raisons de facilité. La figure n'implique en aucune façon des
interactions
 -
- �
spécifiques.
�
spécifiques.
 |
Aucune donnée concernant le repliement
tertiaire n'est incluse, puisqu'aucune interaction
 -
- n'est connue.
n'est connue.
Premièrement, sur la base d'images de microscopie électronique, on peut
localiser la MIR à l'extrémité distale du récepteur, par rapport
à la membrane [22]. En conséquence, E2 et E3 sont
également placés au sommet du repliement. E11 est probablement proche
de la membrane puisqu'il est adjacent à MI (voir plus bas la définition de
position des segments transmembranaires). Ensuite, on peut faire
l'hypothèse que chaque segment d'au moins 4 résidus totalement prédits
exposés au solvant fait saillie à la surface de la sous-unité. Cette
contrainte implique un coude de la structure 1D entre E5 et E6,
E8 et E9, E9 et E10, E10 et E11. Les débuts de
E7 et de E8 sont liés par un pont di-sulfure, et sont donc très
proches l'un de l'autre. Ce pont di-sulfure force un nouveau coude entre
E7 et E8. Cette «Cys-loop» est la partie la plus
conservée du domaine amino-terminal des sous-unités de LGIC. Bien
que la moitié n'en soit pas prédite dans une structure périodique, on peut
raisonnablement penser que la région entière adopte une conformation
fortement contrainte. Finalement, un coude est introduit entre E3 et
E5 afin de respecter la taille observée de la sous-unité qui saille de
60 Å à partir de la membrane, avec le plus grande axe de la section
transversale d'approximativement 40 Å (voir la figure 10.7).
HA
et HB
sont placées perpendiculairement à la membrane afin
de correspondre aux images de microscopie électronique [325],
mais il y a peu de données permettant de contraindre le domaine
HAE1HB.
Cette représentation est pleinement compatible
avec le corpus de données concernant le site de liaison de l'ACh. En
effet, le marquage par affinité et la mutagénèse dirigée ont permis
l'identification d'acides aminés (voir le tableau 7.1) qui
sont distribués à l'interface des sous-unités sur six éléments différents
connus comme A ( 7W8510.2 et
7W8510.2 et
 7Y92,
7Y92,  1W86 et
1W86 et  1Y93), B (
1Y93), B ( 7W148 et
7W148 et
 7Y150,
7Y150,  1W149 et
1W149 et  1Y151), C (
1Y151), C ( 7Y187,
7Y187,
 7C189,
7C189,  7C190 et
7C190 et  7Y194,
7Y194,  1Y190,
1Y190,
 1C192,
1C192,  1C193 et
1C193 et  1Y198) pour le composant
«principal», et D (
1Y198) pour le composant
«principal», et D ( 7W54), E (
7W54), E ( 7L108,
7L108,
 7N110 et
7N110 et  7Q116), et F (
7Q116), et F ( 7D163 et
7D163 et
 7E172) pour le composant «complémentaire». Un autre
résidu a récemment été identifié sur le composant complémentaire
(
7E172) pour le composant «complémentaire». Un autre
résidu a récemment été identifié sur le composant complémentaire
( K34 de souris) [303,261]. Comme il est
localisé dans E1, sa position n'ajoute pas de contraintes
supplémentaires à la représentation 2D (bien que contraignant
peut-être le repliement tertiaire). Les expériences de marquage
par affinité avec des dérivés de toxine ont assigné les
composantes principale et complémentaire aux deux faces des
sous-unités [206]. Cela permet d'orienter la
sous-unité toute entière.
La partie amino-terminale de chaque sous-unité peut être
artificiellement subdivisée en deux domaines. L'un est formé par
HA,
E1 et HB ; l'autre est formé par E2-11.
Cependant, le repliement relatif de ces deux structures reste
hypothétique. La partie HE1HB
doit se replier
sur les feuillets E2-11 afin de former une structure
compacte, contenue dans une surface de 40x60 Å2,
et afin
de rendre compte de la possible contribution des résidus
homologues au résidu
K34 de souris) [303,261]. Comme il est
localisé dans E1, sa position n'ajoute pas de contraintes
supplémentaires à la représentation 2D (bien que contraignant
peut-être le repliement tertiaire). Les expériences de marquage
par affinité avec des dérivés de toxine ont assigné les
composantes principale et complémentaire aux deux faces des
sous-unités [206]. Cela permet d'orienter la
sous-unité toute entière.
La partie amino-terminale de chaque sous-unité peut être
artificiellement subdivisée en deux domaines. L'un est formé par
HA,
E1 et HB ; l'autre est formé par E2-11.
Cependant, le repliement relatif de ces deux structures reste
hypothétique. La partie HE1HB
doit se replier
sur les feuillets E2-11 afin de former une structure
compacte, contenue dans une surface de 40x60 Å2,
et afin
de rendre compte de la possible contribution des résidus
homologues au résidu  K34 de souris au site actif.
K34 de souris au site actif.
PHDhtm [272,271] a été utilisé pour
explorer l'organisation des segments transmembranaires.
PHDhtm est le seul programme de son type qui ne prédit
pas le peptide signal comme étant transmembranaire, probablement à
cause de son manque de conservation. De plus, il prédit les quatre
segments transmembranaires pour tous les membres de la superfamille. SSPCA
fournit le consensus de la sortie de PHDhtm appliqué à
chaque séquence d'AL1. Les résultats, présentés dans le tableau
10.3 montre bien quatre segments.
Tableau 10.3:
Détermination de la position des segments
transmembranaires. Note: les paramètres par défaut ont été
utilisés pour tous les programmes suivants. Un usage un peu plus
réfléchi améliorerait probablement la justesse moyenne des
prédictions. La numérotation est celle d'AL1 (entre parenthèses se
trouve la correspondance avec le peptide mature
adéquat).
| méthode |
|
MI |
MII |
MIII |
MIV |
Motif original
(présenté dans [245] pour  7 et
[258] pour 7 et
[258] pour  1) 1) |
 7gg 7gg |
283-307 (208-232) |
317-337 (238-258) |
353-374 (274-295) |
651-675 (446-470) |
 1tc 1tc |
281-308 (210-236) |
310-339 (239-265) |
348-375 (273-300) |
650-677 (407-433) |
DAS
http://www.biokemi.su.se/ server/DAS/
[73] |
 7gg 7gg |
287-309 (212-234) |
320-340 (241-261) |
352-373 (273-294) |
651-672 (446-467) |
 1tc 1tc |
281-307 (210-235) |
319-339 (245-265) |
349-372 (274-297) |
647-671 (404-427) |
TMPRED
http://ulrec3.unil.ch/software/TMPRED_form.html
[159] |
 7gg 7gg |
290-309 (215-234) |
321-340 (242-261) |
354-372 (275-293) |
649-673 (448-468) |
 1tc 1tc |
281-307 (210-235) |
319-338 (245-264) |
353-371 (278-296) |
652-672 (409-428) |
TOPPRED2
http://www.biokemi.su.se/ server/toppred2/
[333] |
 7gg 7gg |
289-309 (214-234) |
319-339 (240-260) |
353-373 (274-294) |
649-673 (448-468) |
�
 1tc 1tc |
288-309 (217-237) |
317-337 (243-263) |
352-372 (277-297) |
651-672 (408-428) |
SOSUI
http://www.tuat.ac.jp/ mitaku/adv_sosui
[158] |
 7gg 7gg |
286-308 (211-233) |
319-341 (240-262) |
351-372 (272-293) |
650-672 (445-467) |
�
 1tc 1tc |
284-307 (213-235) |
319-341 (245-267) |
349-371 (274-296) |
649-672 (406-428) |
PHDhtm
[271] |
 7gg 7gg |
285-302 (210-227) |
322-339 (243-260) |
351-368 (272-289) |
647-668 (446-463) |
 1tc 1tc |
284-303 (213-231) |
320-338 (246-264) |
350-368 (275-293) |
650-668 (407-424) |
| SSPCA consensus (on each AL1 member) |
 7gg 7gg |
285-303 (210-228) |
322-339 (243-260) |
349-368 (270-289) |
651-668 (446-463) |
 1tc 1tc |
285-303 (214-231) |
322-339 (248-265) |
349-368 (274-393) |
651-668 (408-424) |
A des fins de comparaison, quatre autres programmes ont été
également utilisés sur  1 et
1 et  7. Chacun d'entre eux
prédit les quatre segments transmembranaires des séquences de AL1,
bien que d'autres parties de la sous-unité
soient parfois incorrectement prédites comme transmembranaires. Les résultats
varient avec la méthode mais aussi avec les séquences
utilisées, ce qui montre l'importance d'utiliser le consensus de
multiples analyses. Les longueurs des segments consensus sont 18
pour MI, 17 pour MII, 19 pour MIII et 17 pour MIV. La longueur
des segments consensus est moindre que celle décrite dans les
propositions usuelles [258]. Cependant, cela pourrait
être un artefact dû à l'attitude prudente de l'algorithme de
PHDhtm. SSPCA prédit que le domaine
transmembranaire se replie en une structure mixte
hélice-
7. Chacun d'entre eux
prédit les quatre segments transmembranaires des séquences de AL1,
bien que d'autres parties de la sous-unité
soient parfois incorrectement prédites comme transmembranaires. Les résultats
varient avec la méthode mais aussi avec les séquences
utilisées, ce qui montre l'importance d'utiliser le consensus de
multiples analyses. Les longueurs des segments consensus sont 18
pour MI, 17 pour MII, 19 pour MIII et 17 pour MIV. La longueur
des segments consensus est moindre que celle décrite dans les
propositions usuelles [258]. Cependant, cela pourrait
être un artefact dû à l'attitude prudente de l'algorithme de
PHDhtm. SSPCA prédit que le domaine
transmembranaire se replie en une structure mixte
hélice- /brin-
/brin- ,
à peu près sans résidu
non-structuré. La figure 10.5 montre une tentative de
représentation en 2D. Cependant, comme aucune information n'existe
à propos de l'orientation précise des structures dans la membrane,
les angles représentés sont arbitraires, excepté dans le cas de
l'hélice présente dans le segment MII qu'on sait être plus ou
moins perpendiculaire à la membrane. De plus, la longueur des
structures prédites est peu précise.
Mises à part HF
et HG,
le domaine cytoplasmique est prédit comme
totalement accessible au solvant et dans une structure apériodique.
,
à peu près sans résidu
non-structuré. La figure 10.5 montre une tentative de
représentation en 2D. Cependant, comme aucune information n'existe
à propos de l'orientation précise des structures dans la membrane,
les angles représentés sont arbitraires, excepté dans le cas de
l'hélice présente dans le segment MII qu'on sait être plus ou
moins perpendiculaire à la membrane. De plus, la longueur des
structures prédites est peu précise.
Mises à part HF
et HG,
le domaine cytoplasmique est prédit comme
totalement accessible au solvant et dans une structure apériodique.
10.4 Discussion
Des travaux antérieurs ont montré que la justesse des prédictions de
structure secondaire est accrue avec la combinaison de plusieurs
algorithmes indépendants [235,27,368]. Afin de
réaliser la meilleure prédiction disponible des sous-unités de nAChR,
j'ai intégré les résultats de plusieurs programmes de 3ème
génération, utilisant l'information venant d'alignements multiples. Ces
programmes ont été sélectionnés sur la base de leur efficacité sur des
ensembles de protéines tests, aux structures secondaires connues
[275] ou durant des prédictions en aveugle
[270,171]. De plus, chaque programme à été appliqué à
chaque séquence des alignements, afin d'augmenter le rapport signal/bruit.
Deux principales prédictions ab initio du nAChR ont été
rapportées durant les deux dernières décades. FINER-MOORE
et STROUD [105] utilisaient l'algorithme du
GOR [124] pour les régions (supposées)
extra-membranaires, et une analyse (par transformée de Fourier) de
la périodicité de l'hydrophobie pour les régions transmembranaires
putatives. Récemment ORTELLS [243] a
présenté une prédiction de structure secondaire basée sur une
approche ressemblant à l'algorithme de CHOU et
FASMAN [51]. La principale différence entre la
méthode initiale et celle utilisée dans [243] réside
dans la définition des initiateurs des structures secondaires. Au
lieu de les prédire seulement par la séquence (via des tableaux
statistiques) comme dans CHOU et FASMAN, les
initiateurs étaient déterminés comme suit: Un initiateur est
défini comme un résidu constamment prédit dans le même état, à
travers plusieurs ensembles de sous-unités de LGIC,
analysés par des algorithmes de 1ère
et de 2ème
génération. Une autre différence réside dans le fait que la
propagation à partir des initiateurs est unidirectionnelle dans
[243], dans le sens amino-terminal vers
carboxy-terminal, alors qu'elle est bi-directionnelle dans
[51].
La justesse de prédiction moyenne a déjà été discutée ailleurs
(voir [167] et [234] pour des
évaluations initiales et [275,277] pour des
revues récentes), mais la différence entre ces premiers travaux et
la prédiction présentée ici, pourrait atteindre 20 %. En effet
sur un ensemble test identique, l'algorithme de CHOU et
FASMAN atteignit 49 % alors que PHD atteignait
72,5 % [275].
Tableau 10.4 :
Contenu en hélice- et
brin-
et
brin- mesuré et prédit dans une sous-unité entière. Note:
[41] est clairement à part, diminuant le contenu
moyen en hélice et augmentant le contenu moyen en
brin.
mesuré et prédit dans une sous-unité entière. Note:
[41] est clairement à part, diminuant le contenu
moyen en hélice et augmentant le contenu moyen en
brin.
| |
contenu en hélice |
contenu en brin |
rapport |
| [360] |
39 % |
33 % |
1,18 |
| [41] |
18,7 % |
42 % |
0,45 |
| [223] |
39 % |
35 % |
1,11 |
| [346] |
48 % |
26 % |
1,85 |
| moyenne des valeurs expérimentales |
36,2 % |
34 % |
1,15 |
| [105] |
44 % |
27 % |
1,63 |
| [243] |
29,7 % |
24,9 % |
1,19 |
| consensus SSPCA |
25,8 % |
22,3 % |
1,16 |
| consensus affiné |
24,2 % |
22,5 % |
1,07 |
Le contenu en hélice- et en brin-
et en brin- du nAChR entier a été
mesuré par plusieurs groupes utilisant des méthodes de mesures
spectroscopiques différentes
[220,360,41,223]. Les résultats montrent
une forte variabilité qui ne peut être seulement due aux différences dans
l'environnement du récepteur (lipides, détergent etc.). En effet, la
mesure du contenu en hélice varie de 18,7 % [41] à 48 %
[346], la mesure du contenu en brin (sans les
du nAChR entier a été
mesuré par plusieurs groupes utilisant des méthodes de mesures
spectroscopiques différentes
[220,360,41,223]. Les résultats montrent
une forte variabilité qui ne peut être seulement due aux différences dans
l'environnement du récepteur (lipides, détergent etc.). En effet, la
mesure du contenu en hélice varie de 18,7 % [41] à 48 %
[346], la mesure du contenu en brin (sans les
 -turn) variant elle de 26 % [346] à 42 %
[41], et le rapport calculé hélice/brin variant de 0,45
[41] à 1,85 [346], [223,360]
trouvant des valeurs intermédiaires 1,11 et 1,18 (tableau
10.4).
Le consensus de SSPCA fournit des valeurs légèrement plus
faibles que la moyenne des expériences en contenu d'hélice et de
brin, bien que le rapport soit à peu près identique. Dans la
partie amino-terminale (selon la définition de [346] et
non la détermination des segments transmembranaires présentée
ici), notre prédiction donne un contenu en hélice-
-turn) variant elle de 26 % [346] à 42 %
[41], et le rapport calculé hélice/brin variant de 0,45
[41] à 1,85 [346], [223,360]
trouvant des valeurs intermédiaires 1,11 et 1,18 (tableau
10.4).
Le consensus de SSPCA fournit des valeurs légèrement plus
faibles que la moyenne des expériences en contenu d'hélice et de
brin, bien que le rapport soit à peu près identique. Dans la
partie amino-terminale (selon la définition de [346] et
non la détermination des segments transmembranaires présentée
ici), notre prédiction donne un contenu en hélice- équivalent (13,7 % contre 12 %) et moins de brin-
équivalent (13,7 % contre 12 %) et moins de brin- (31,7 % contre 51 %) que ce qui est observé dans l'unique étude
existante [346].
(31,7 % contre 51 %) que ce qui est observé dans l'unique étude
existante [346].
Figure 10.6 :
A-Comparaison de
notre prédiction de structure secondaire avec celles de
[140,322,243]. Toutes ces prédictions
sont projetées sur la partie amino-terminale d' 1 de
torpille. B-Comparaison de notre prédiction de structure
secondaire avec celle de [245] basée sur l'analogie
avec l'entérotoxine. Les prédictions sont projetées sur la partie
transmembranaire d'
1 de
torpille. B-Comparaison de notre prédiction de structure
secondaire avec celle de [245] basée sur l'analogie
avec l'entérotoxine. Les prédictions sont projetées sur la partie
transmembranaire d' 7 de poulet.
7 de poulet.
 |
Au niveau du domaine extracellulaire amino-terminal, toutes les
approches prédisent une structure principalement repliée en
brin- [figure 10.6A]. Cependant la position
des structures, comme leur nombre, diffère considérablement entre
les différentes études. Le fort contenu en
[figure 10.6A]. Cependant la position
des structures, comme leur nombre, diffère considérablement entre
les différentes études. Le fort contenu en  est aussi
cohérent avec les images de microscopie électronique (bien que
trois hélices aient été proposées dans ces dernières
investigations) [325,326]. Les structures
prédites par ORTELLS [243] sont plus longues que celles
présentées dans l'étude présente et plus longues que les valeurs
observées dans la PDB. Les deux larges hélices prédites
dans la moitié amino-terminale de la partie extracellulaire ont
une longueur de 20 résidus alors que nos prédictions sont de 12 et 14
résidus et la moyenne de la PDB est de 9. De même la longueur
moyenne des brins-
est aussi
cohérent avec les images de microscopie électronique (bien que
trois hélices aient été proposées dans ces dernières
investigations) [325,326]. Les structures
prédites par ORTELLS [243] sont plus longues que celles
présentées dans l'étude présente et plus longues que les valeurs
observées dans la PDB. Les deux larges hélices prédites
dans la moitié amino-terminale de la partie extracellulaire ont
une longueur de 20 résidus alors que nos prédictions sont de 12 et 14
résidus et la moyenne de la PDB est de 9. De même la longueur
moyenne des brins- est de 7,2 dans [243], 5,8
dans l'étude présente, et 5,1 dans la PDB. Ces désaccords
sont probablement dus à la méthode que ORTELLS utilise pour
propager les éléments structuraux. Une fois initié, chaque élément
est étendu en avant jusqu'à ce qu'il atteigne un initiateur
différent ou une proline ou une glycine. La figure
10.6A fournit également une comparaison avec les
structures secondaires dérivées par les méthodes de fold
recognition [140,322]. Dans ce cas, non
seulement les longueurs des motifs, mais aussi leurs positions
dans la séquence sont très différentes.
La localisation des quatre segments transmembranaires putatifs a été
originellement réalisée par analyse de diagrammes d'hydropathie. Cette
méthode, bien que d'un grand intérêt et facile à utiliser, ne s'applique
pas de manière satisfaisante dans le cas des canaux transmembranaires. En
effet, les résidus longeant le pore ionique dans l'état ouvert ne sont pas
supposés être particulièrement hydrophobes. De plus, dans une protéine
contenant plusieurs domaines transmembranaires, comme le nAChR, les
segments internes peuvent être isolés de l'environnement lipidique. Enfin,
des séries d'acides aminés hydrophobes peuvent être externes à la membrane
(par exemple, enfouis au coeur de la protéine). Comme conséquence,
certains segments transmembranaires peuvent avoir été mal prédits. Par
exemple, pour la sous-unité
est de 7,2 dans [243], 5,8
dans l'étude présente, et 5,1 dans la PDB. Ces désaccords
sont probablement dus à la méthode que ORTELLS utilise pour
propager les éléments structuraux. Une fois initié, chaque élément
est étendu en avant jusqu'à ce qu'il atteigne un initiateur
différent ou une proline ou une glycine. La figure
10.6A fournit également une comparaison avec les
structures secondaires dérivées par les méthodes de fold
recognition [140,322]. Dans ce cas, non
seulement les longueurs des motifs, mais aussi leurs positions
dans la séquence sont très différentes.
La localisation des quatre segments transmembranaires putatifs a été
originellement réalisée par analyse de diagrammes d'hydropathie. Cette
méthode, bien que d'un grand intérêt et facile à utiliser, ne s'applique
pas de manière satisfaisante dans le cas des canaux transmembranaires. En
effet, les résidus longeant le pore ionique dans l'état ouvert ne sont pas
supposés être particulièrement hydrophobes. De plus, dans une protéine
contenant plusieurs domaines transmembranaires, comme le nAChR, les
segments internes peuvent être isolés de l'environnement lipidique. Enfin,
des séries d'acides aminés hydrophobes peuvent être externes à la membrane
(par exemple, enfouis au coeur de la protéine). Comme conséquence,
certains segments transmembranaires peuvent avoir été mal prédits. Par
exemple, pour la sous-unité  1 du récepteur glycine de rat, le
programme SOSUI [216], basé sur les propriétés
physique des acides aminés, ne prédit pas les segments MII et MIV comme
unités transmembranaires, non plus que le programme TMPRED
[159], basé sur la comparaison avec une banque de donnée de
segments transmembranaires connus. Les prédictions originelles varient
d'un auteur à l'autre puisque l'on trouve (pour MI-MIII de
1 du récepteur glycine de rat, le
programme SOSUI [216], basé sur les propriétés
physique des acides aminés, ne prédit pas les segments MII et MIV comme
unités transmembranaires, non plus que le programme TMPRED
[159], basé sur la comparaison avec une banque de donnée de
segments transmembranaires connus. Les prédictions originelles varient
d'un auteur à l'autre puisque l'on trouve (pour MI-MIII de
 1tc�10.3):
1tc�10.3):
| Séquence |
RIPLYFVVNVIIPCLLFSFLTVLVFYLPTDSGEKMTLSISVLLSLTVFLLVIVELIPSTSSAVPLIGKYMLFTMIFVISSIIVTVVVINTHHR |
| [58] |
...TTTTTTTTTTTTTTTTTTTTTTTT.......TTTTTTTTTTTTTTTTTTT................TTTTTTTTTTTTTTTTTTT..... |
| [87] |
.TTTTTTTTTTTTTTTTTTTTTTTTTTT..TTTTTTTTTTTTTTTTTTTTTTTTTT........TTTTTTTTTTTTTTTTTTTTTTTTTTTT. |
| [236] |
..TTTTTTTTTTTTTTTTTTTTTTTTTT......TTTTTTTTTTTTTTTTTTT...............TTTTTTTTTTTTTTTTTTTTTT... |
| [231] |
.TTTTTTTTTTTTTTTTTTTTTTTTTTTTT...TTTTTTTTTTTTTTTTTTTTTT.............TTTTTTTTTTTTTTTTTTTTTTT.. |
La position des segments transmembranaires est prédite par PHDhtm
à 95 % de justesse. Cette précision est supérieure à l'incertitude
présentée ci-dessus.
Alors que les prédictions de la position des segments
transmembranaires peut apparaître fiable, l'assignation
structurale doit néanmoins être acceptée avec prudence.
Puisqu'aucun des programmes utilisés dans ce travail n'a été
entraîné sur des protéines membranaires, la justesse espérée dans
les prédictions transmembranaires est probablement plus basse que
celle des régions extramembranaires. En effet, l'environnement
lipidique impose des contraintes sur la structure10.4. Cependant, j'ai mené un test des programmes
utilisés dans la présente étude sur un ensemble test de protéines
de structure connue [Tableau 10.5]. Le groupe test
était composé de 20 chaînes ne comportant pas d'identité par paire
supérieure à 25 % et dont la résolution était supérieure à
3,5 Å.
Tableau 10.5 :
Protéines transmembranaires utilisées pour tester les
programmes de prédiction
| Identificateur PDB |
Fonction |
Espèce |
Résolution |
| 1AFO |
Glycophorin A |
Homo sapiens |
RMN |
| 1AIJ_H |
Photosynthetic reaction center |
Rhodobacter sphaeroides |
2.2 Å |
| 1ATY |
F1F0 ATP synthase |
Escherichia coli |
RMN |
| 1AUW |
Delta2
crystallin |
Anas platyrhynchos |
2.5 Å |
| 1CIY |
Delta endotoxin cryia(A) |
Bacillus
thuringiensis |
2.25 Å |
| 1COL |
Colicin A |
Escherichia coli |
2.4 Å |
| 1HTM |
Hemagglutinin |
virus de Influenza |
2.5 Å |
| 1KSA |
Bacteriochlorophyll A |
Chlorobium
tepidum |
2.5 Å |
| 1KZU_A |
Light harvesting
complex |
Rhodopseudomonas acidophila |
2.5 Å |
| 1KZU_B |
idem |
idem |
idem |
| 1MAL |
Maltoporin |
Eschericha coli |
3.1 Å |
| 1MDT |
Monomeric diphteria toxin |
Corynebacterium diphteriae |
2.3 Å |
| 1PRC_C |
Photosynthetic reaction center |
Rhodopseudomonas viridis |
2.3 Å |
| 1PRC_M |
idem |
idem |
idem |
| 1PRN |
Porin |
Rhodopseudomonas
blastica |
1.96 Å |
| 1VMO |
Vitelline membrane
protein I |
Gallus gallus |
2.2 Å |
| 2BRD |
Bacteriorhodopsin |
Halobacterium halobium |
3.5 Å |
| 2OMF |
Ompf porin |
Escherichi coli |
2.4 Å |
| 2POR |
Porin |
Rhodopseudomonas viridis |
1.8 Å |
| 7AHL |
Alpha hemolysin |
Staphylococcus |
1.9 Å |
Les critères quantitatifs utilisés dans le tableau
10.6 pour quantifier les résultats sont les
suivants:
Pi est le nombre de résidus prédits dans l'état i effectivement dans
l'état i et Oi est le nombre de résidus prédits dans l'état i
n'étant pas dans l'état i (Overpredicted). Pi+Oi représente
donc le nombre total de résidus prédits dans l'état i.
Qipred est
donc un indice de fiabilité (mais non de justesse).
Ui est le nombre de résidus dans l'état i n'ayant pas été prédits
(Underpredicted). Pi+Ui représente donc le nombre de résidus
réellement dans l'état i. Qiobs est donc un indice de justesse
(mais non de fiabilité).
Où T est le nombre total de résidus. Q3 représente donc la
justesse globale.
La valeur 0 représente la prédiction aléatoire, 1 étant la
prédiction parfaite.
Tableau 10.6:
Résultats
détaillés de chaque programme de prédiction sur l'ensemble test de
protéines transmembranaires. En italique est inscrite la justesse observée
sur des ensembles tests de protéines solubles. Les nombres sont ceux
annoncés par les auteurs mais correspondent globalement à ce qu'on observe
sur des tests en aveugle. Cependant PHD est le seul atteignant
réellement les 72 %, PREDATOR étant quant à lui très
surestimé.
| programme |
Q3 |
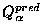 |
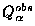 |
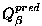 |
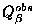 |
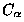 |
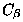 |
| PHD |
67,3 |
76,3 |
70,1 |
65,8 |
55,1 |
0,6 |
0,45 |
| [275] |
71,6 |
60
|
57 |
63 |
62 |
0,61
|
0,52 |
| DSC |
64,6 |
77,6 |
62,5 |
64,1 |
53,9 |
0,57 |
0,43 |
| [171] |
70,1 |
|
73,5 |
|
64,9 |
0,58 |
0,51 |
| NNSSP |
61,8 |
78,4 |
68 |
69,1 |
30,7 |
0,58 |
0,34 |
| [287] |
72,2 |
76,2 |
72,4
|
67,4 |
52,2 |
0,64 |
0,50 |
| PREDATOR |
60,5 |
72,4 |
60,7 |
57,4 |
37,3 |
0,51 |
0,30 |
| [114] |
74,8
|
|
|
|
|
0,61 |
0,44
|
Sur 5321 résidus, 1804 ont été prédits en hélice- et 1584 en
brin-
et 1584 en
brin- . La justesse observée était 67,3 % pour PHD,
64,6 % pour DSC, 61,8 % pour NNSSP et 60,5 % pour
PREDATOR. Ces valeurs sont plus basses que celles déterminées
pour les protéines globulaires (de 70 % à 75 %) mais sont bien
meilleures que des valeurs aléatoires et supérieures à celles des
algorithmes de prédiction de 1ère génération dans le cas des
protéines globulaires solubles. Les prédictions des hélice-
. La justesse observée était 67,3 % pour PHD,
64,6 % pour DSC, 61,8 % pour NNSSP et 60,5 % pour
PREDATOR. Ces valeurs sont plus basses que celles déterminées
pour les protéines globulaires (de 70 % à 75 %) mais sont bien
meilleures que des valeurs aléatoires et supérieures à celles des
algorithmes de prédiction de 1ère génération dans le cas des
protéines globulaires solubles. Les prédictions des hélice- sont
meilleures que celles des brin-
sont
meilleures que celles des brin- , l'erreur la plus commune étant la
sous-prédiction des brin-
, l'erreur la plus commune étant la
sous-prédiction des brin- .
.
Des prédictions structurales ont déjà été faites pour le domaine
transmembranaire, basées sur des arguments analogiques. UNWIN
[325] suggéra, sur la base de ses images de microscopie
électronique, que la région transmembranaire du nAChR pourrait avoir un
repliement similaire à celui de certains domaines des entérotoxines.
ORTELLS et LUNT[245] exploitèrent cette
idée pour modéliser une partie de la région transmembranaire des
LGIC en utilisant comme structure de départ la résolution
cristallographique du domaine B de l'entérotoxine thermo-sensible de
Escherichia coli [304]. Le modèle en résultant présente
une structure secondaire mixte  /
/ , où MII est
tout-
, où MII est
tout- , MI est tout-
, MI est tout- et MIII est
et MIII est  /
/ [figure
4.6B], la région
[figure
4.6B], la région  -hélicale de MIII ayant été ajoutée
a posteriori au modèle. Plusieurs remarques peuvent être faites à
propos de cette étude, mis-à-part le fait que le gabarit n'ai jamais été
trouvé jusqu'à maintenant par aucun programme de fold recognition.
Premièrement, l'entérotoxine n'est pas une protéine intégrale de membrane,
et n'est donc pas un gabarit adéquat pour le domaine intramembranaire du
nAChR. ORTELLS et LUNT [245] ont enlevé le
premier brin, qui interagit avec le cinquième. La structure en résultant
pourrait être moins stable, l'un des feuillets n'étant plus composé que de
deux brins anti-parallèles. Comme le précisent les auteurs, l'addition
ultérieure de MIII et MIV, modelés en hélices (partiellement pour MIII)
peut résulter en une ségrégation de la partie «entérotoxine» des
lipides. Les prédictions de structure secondaire présentées ici ne
correspondent pas avec celles proposées par[245] [figure
10.6B). Une comparaison à trois états donne seulement 33 %
de résidus identiquement prédits.
-hélicale de MIII ayant été ajoutée
a posteriori au modèle. Plusieurs remarques peuvent être faites à
propos de cette étude, mis-à-part le fait que le gabarit n'ai jamais été
trouvé jusqu'à maintenant par aucun programme de fold recognition.
Premièrement, l'entérotoxine n'est pas une protéine intégrale de membrane,
et n'est donc pas un gabarit adéquat pour le domaine intramembranaire du
nAChR. ORTELLS et LUNT [245] ont enlevé le
premier brin, qui interagit avec le cinquième. La structure en résultant
pourrait être moins stable, l'un des feuillets n'étant plus composé que de
deux brins anti-parallèles. Comme le précisent les auteurs, l'addition
ultérieure de MIII et MIV, modelés en hélices (partiellement pour MIII)
peut résulter en une ségrégation de la partie «entérotoxine» des
lipides. Les prédictions de structure secondaire présentées ici ne
correspondent pas avec celles proposées par[245] [figure
10.6B). Une comparaison à trois états donne seulement 33 %
de résidus identiquement prédits.
La représentation 2D rend compte de l'information élémentaire
concernant le site de liaison des ligands compétitifs. Les
prédictions de structure secondaire suggèrent que les éléments B,
F et C sont portés par des segments sans structure périodique,
alors que les éléments A, E et D sont, au moins en partie, portés
par des segments structurés.
Au niveau du composant complémentaire, 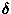 W57 (AL1104),
marqué par affinité, est localisé au centre de HB. Des
mutations à la position AL1106 modulent à la
fois la pharmacologie des agonistes et celle des antagonistes
[67,150,49]. Cependant, les chaînes
latérales des résidus AL1102 et AL1104 pointent vers
l'extérieur à partir des faces opposées de l'hélice, impliquant
que AL1104 transmet ses effets indirectement, peut-être par
des altérations locales de la structure.
W57 (AL1104),
marqué par affinité, est localisé au centre de HB. Des
mutations à la position AL1106 modulent à la
fois la pharmacologie des agonistes et celle des antagonistes
[67,150,49]. Cependant, les chaînes
latérales des résidus AL1102 et AL1104 pointent vers
l'extérieur à partir des faces opposées de l'hélice, impliquant
que AL1104 transmet ses effets indirectement, peut-être par
des altérations locales de la structure.
Au niveau de l'élément E, deux brins- successifs sont
prédis, E5 portant les résidus marqués
successifs sont
prédis, E5 portant les résidus marqués 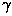 L109
(AL1160) et
L109
(AL1160) et  Y111 (AL1162) et E6 portant
Y111 (AL1162) et E6 portant
 Y117 (AL1175) [302,303]. Une
possibilité serait que les brins-
Y117 (AL1175) [302,303]. Une
possibilité serait que les brins- interagissent dans un
feuillet anti-parallèle, qui dirigerait les chaînes latérales des
résidus marqués dans la même direction.
interagissent dans un
feuillet anti-parallèle, qui dirigerait les chaînes latérales des
résidus marqués dans la même direction.
Finalement, le segment portant l'élément F contient le site de
liaison du calcium responsable de la potentiation de l'action des
agonistes [121].
Au niveau du composant principal, les expériences de mutagénèse
ont montré que plusieurs mutations, localisées dans le voisinage
des résidus des éléments B et C marqués par affinité, altèrent
profondément les propriétés pharmacologiques (région
AL1210-AL1214, et AL1256-AL1259)
[68]. Comme ces régions sont prédites sans
structures secondaires régulières, les mutations pourraient
altérer la liaison indirectement, par des réorganisations des
boucles.
Chaque segment transmembranaire du récepteur est prédit comme une
structure mixte  /
/ . Cette prédiction doit être prise avec
précaution, comme noté au-dessus. De plus, des expériences de marquage par
affinité avec une sonde hydrophobe radioactive supporte une organisation
en hélice-
. Cette prédiction doit être prise avec
précaution, comme noté au-dessus. De plus, des expériences de marquage par
affinité avec une sonde hydrophobe radioactive supporte une organisation
en hélice- des segments MIII et MIV [28]. MIII est
prédit en hélice-
des segments MIII et MIV [28]. MIII est
prédit en hélice- jusqu'à AL1362 (
jusqu'à AL1362 ( 7F283) alors que
HE�atteint seulement AL1355 (
7F283) alors que
HE�atteint seulement AL1355 ( 7S276) et MIV est prédit
en hélice-
7S276) et MIV est prédit
en hélice- jusqu'à AL1668 (
jusqu'à AL1668 ( 7I463), alors que
HH� atteint seulement AL1657 (
7I463), alors que
HH� atteint seulement AL1657 ( 7F452). En ce qui
concerne MII, connu pour faire face à la lumière du canal ionique, les
présentes prédictions pourraient mener à une re-considération de
l'architecture couramment acceptée du mécanisme de passage des ions. La
prédiction de MII commence au niveau de l'acide aminé AL1323, 4
résidus après la vision du modèle standard. De plus l'hélice MII est
prédite un peu plus courte. La plus grande partie des données de marquage
par affinité et de mutagénèse dirigée est bien représentée par une
structure hélicoïdale [265,3]. D'un autre côté, des
résultats récents [351] supportent une structure allongée pour
le court segment allant du résidu AL1310 (
7F452). En ce qui
concerne MII, connu pour faire face à la lumière du canal ionique, les
présentes prédictions pourraient mener à une re-considération de
l'architecture couramment acceptée du mécanisme de passage des ions. La
prédiction de MII commence au niveau de l'acide aminé AL1323, 4
résidus après la vision du modèle standard. De plus l'hélice MII est
prédite un peu plus courte. La plus grande partie des données de marquage
par affinité et de mutagénèse dirigée est bien représentée par une
structure hélicoïdale [265,3]. D'un autre côté, des
résultats récents [351] supportent une structure allongée pour
le court segment allant du résidu AL1310 ( 7S235) au résidu
AL1319 (
7S235) au résidu
AL1319 ( 7S240). De plus, MI et MII semblent proches l'un de
l'autre [2]. En conséquence, la portion cytoplasmique liant
MI et MII est prédite plus longue, et pourrait former une
7S240). De plus, MI et MII semblent proches l'un de
l'autre [2]. En conséquence, la portion cytoplasmique liant
MI et MII est prédite plus longue, et pourrait former une
 -hairpin (E13-E14). La longueur de la boucle
liant les brins est variable selon la sous-unité considérée. Des
expériences récentes de mutagénèse réalisées dans le laboratoire mettent
en évidence une contribution majeure de la portion centrale de cette
boucle cytoplasmique en tant que filtre de sélectivité du canal ionique.
En outre, sa conformation, plus que sa séquence précise, aurait un effet
critique sur les propriétés de sélectivité du canal (Corringer et coll., en
préparation).
-hairpin (E13-E14). La longueur de la boucle
liant les brins est variable selon la sous-unité considérée. Des
expériences récentes de mutagénèse réalisées dans le laboratoire mettent
en évidence une contribution majeure de la portion centrale de cette
boucle cytoplasmique en tant que filtre de sélectivité du canal ionique.
En outre, sa conformation, plus que sa séquence précise, aurait un effet
critique sur les propriétés de sélectivité du canal (Corringer et coll., en
préparation).
HF et HG� sont amphipathiques, et prédites avec une face
exposée au solvant et une face enfouie. Le moment hydrophobe maximum
[96] (déterminé par le programme MOMENT de la
suite WISCONSIN du GCG [85] avec une
fenêtre de 8 résidus) est 0,19 pour HF
� (faible) et 0,57 pour
HG� (fort) . De plus, les deux hélices présentent une signature de
leucine-zipper. Sur 79 séquences:
AL1393: 61 Leucines
AL1400: 62 Leucines, 14 méthionines
AL1611: 30 Leucines , 6 méthionines
une position hydrophobe conservée en AL1615
AL1618: seulement 2 Leucines mais 21 Isoleucines
une position hydrophobe conservée en AL1622
AL1625: 17L, 30M
Ces deux hélices cytoplasmiques pourraient interagir dans
un arrangement coiled-coil, à l'intérieur de la sous-unité ou
même entre sous-unités (HF� d'une sous-unité interagissant avec
HG
� d'une autre). Ce motif pourrait être critique pour le
processus d'oligomérisation. En effet Yu et coll. [365] ont montré que
deux délétions d'acides aminés appartenant à HF
� et HG�
perturbent la formation du pentamère.
Sur la base de la représentation 2D prédite plus haut, on peut proposer un
modèle hypothétique pour l'assemblage de cinq sous-unités au sein d'un
récepteur oligomérique.
Chaque sous-unité est généralement vue comme un bâtonnet vertical,
cinq d'entre eux formant le récepteur [325]. Cependant,
dans les reconstructions en volume effectuées à partir des images
de diffraction électronique, la molécule de récepteur montre une
torsion droite, chaque groupe de densité tournant autour de l'axe
de symétrie [319]. On peut spéculer que la portion
extracellulaire de chaque sous-unité ne présente pas la forme d'un
bâtonnet, mais est plus aplatie [figure 10.7].
Figure 10.7 :
Modèle hypothétique de l'arrangement pentamérique
des sous-unités autour de l'axe de symétrie du récepteur. L'image de
gauche est basée sur la vision classique des sous-unités en bâtonnets.
L'image de droite est basée sur l'idée présentée ici de sous-unités
aplaties. Les courbes noires sont adaptées des lignes d'iso-densité de la
figure 8 de [325]. Les étoiles représentent le site de liaison
de l' -bgt, déterminé dans [177].
-bgt, déterminé dans [177].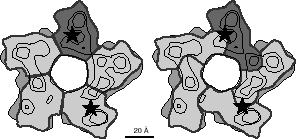 |
���
Son plus grand axe parallèle à la membrane serait de l'ordre de 40 Å et
non de 25 Å. Le site de liaison des ligands compétitifs resterait
localisé entre les trois densités observées par UNWIN
[325]. Mais, alors que dans ce dernier travail elles sont
supposées être des hélices- appartenant à la même sous-unité, ici
les deux hélices restantes sont supposées appartenir à deux sous-unités
différentes. Le site serait situé à l'interface entre deux sous-unités, ce
qui est supporté par de nombreuses données expérimentales. Cette
représentation met l'accent sur l'asymétrie caractéristique de chaque
sous-unité de la super-famille, chaque
«protomère», au sein de l'oligomère symétrique
[217,46].
appartenant à la même sous-unité, ici
les deux hélices restantes sont supposées appartenir à deux sous-unités
différentes. Le site serait situé à l'interface entre deux sous-unités, ce
qui est supporté par de nombreuses données expérimentales. Cette
représentation met l'accent sur l'asymétrie caractéristique de chaque
sous-unité de la super-famille, chaque
«protomère», au sein de l'oligomère symétrique
[217,46].
Nous avons présenté une nouvelle prédiction de la structure
secondaire d'une sous-unité typique de nAChR sur la base de
l'analyse de la séquence primaire, en utilisant la combinaison
d'algorithmes de troisième génération. Ces prédictions pourront
servir de base pour les méthodes de fold recognition. Les
prédictions de topologie transmembranaire, d'accessibilité au
solvant, ainsi que les données expérimentales disponibles, ont été
ajoutées afin de formuler une représentation 2D comprenant un
minimum d'hypothèses non-vérifiées. Cette représentation pourra
aussi servir de cadre de réflexion pour proposer de nouvelles
approches de mutagénèse et de construction de protéines chimères
au sein de la superfamille, afin de relier l'organisation
tri-dimensionnelle du récepteur et ses propriétés physiologiques
et pharmacologiques.
Footnotes
- ...[122]10.1
- Les récepteurs ionotropiques du glutamate des
vertébrés constituent une superfamille séparée, dans laquelle les sites de
liaison des agonistes sont inclus au sein d'une sous-unité et non à
l'interface entre sous-unités [247] cf. chapitre
2
- ...
 7W8510.2
7W8510.2
- les numérotations sont celles de
 7 mature de poulet et
7 mature de poulet et  1 mature de torpille.
1 mature de torpille.
- ...tc10.3
- La sous-unité
 tc
est analysée
dans [58] et les sous-unités
tc
est analysée
dans [58] et les sous-unités 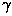 gg�
et
gg�
et
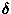 gg
sont analysées dans [231]
gg
sont analysées dans [231]
- ... structure10.4
- La plus
simple étant qu'il n'y a pas de formation de liaison hydrogène
avec l'extérieur. Dans un solvant aqueux, la rupture d'une liaison
hydrogène intra-protéine ne coûte quasiment rien (
 1.7
KJ.mol-1) car elle est remplacée par deux autres avec des
molécules d'eau. En revanche, dans un solvant apolaire, rien ne
vient la remplacer (d'où une perte de
1.7
KJ.mol-1) car elle est remplacée par deux autres avec des
molécules d'eau. En revanche, dans un solvant apolaire, rien ne
vient la remplacer (d'où une perte de
 20
KJ.mol-1)
20
KJ.mol-1)



Next: 11. Théorie et pratique
Up: No Title
Previous: 9. Histoire évolutive des
Nicolas Le Novère
1999-06-19
 et brin-
et brin-
 /
/

![\begin{displaymath}C[P(m_i,s_x),P(m_j,s_y)]= \frac{100}{card(\bar{G})} \sum_{\alpha
\in \bar{G}} \delta [P(m_i,s_x),P(m_j,s_y)]_{\alpha}
\end{displaymath}](img79.gif)
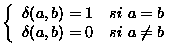 (
(![\begin{displaymath}\mu_{i,j}=\frac{1}{S} \sum_{x=1}^{S}
C[P(m_i,s_x),P(m_j,s_x)]
\end{displaymath}](img82.gif)
![\begin{displaymath}\sigma_{x,y}=\frac{1}{M} \sum_{i=1}^{M} C[P(m_i,s_x),P(m_i,s_y)]
\end{displaymath}](img85.gif)